Im Interventionsraum der Zukunft beginnt der Closed-Loop-Prozess mit der Anwendung und Fusion verschiedener bildgebender Verfahren (3-Tesla-MR-Tomographen, PET-Scanner, CT-Scanner SOMATOM Force) (Leitprojekt M²IBID), die genaue Informationen zur schnellen Navigation der robotergestützten Assistenzsysteme (Artis ZEEGO® System und Leichtbauroboter) im Interventionsraum und der gezielten Biopsieentnahme (Leitprojekt M²INT) liefern. Das Assistenzsystem befördert die Proben direkt zur Bioanalyse mittels Massenspektrometrie, auf die unmittelbar die radiochemische Produktion von Diagnostika und Therapeutika (Leitprojekt M²oBiTE) folgt. Diese Substanzen werden anschließend wieder mit dem Roboter gestützten Assistenzsystem minimalinvasiv appliziert (M²INT). Alle neu entwickelten Methoden werden in den Closed-Loop-Prozess unter optimaler Nutzung aller personellen und infrastrukturellen Ressourcen integriert (Querschnittsprojekte ProM²OLIE und M²PROCIT), so dass ein zeitoptimierter Behandlungspfad für jede Patientin und jeden Patienten von der Aufnahme in der Klinik bis zu Entlassung nach der erfolgten Therapie gewährleistet ist.
Das Projekt M²INT befasst sich mit der Entwicklung eines teilautomatisierten Interventionssystems für die effiziente Durchführung von minimalinvasiven, individualisierten Maßnahmen der Radiologie und der Radiotherapie bei oligometastasierten Patienten. Im Mittelpunkt stand in der ersten Phase die Einbindung von klinischen Verfahren der interventionellen Radiologie, z. B. Biopsie, und der Strahlentherapie, z. B. Brachytherapie, in das Interventionssystem.
Das Ziel der ersten Förderphase war es, die Prozesszeiten bei gesteigerter Präzision so zu verkürzen, dass die Behandlung (Diagnose, Analytik und Therapie) von oligometastasierten Patienten in einem durchgängigen Prozess an höchstens einem Tag durchführbar wird („One-Stop-Shop“) und der erhöhte technische Aufwand wirtschaftlich zu rechtfertigen ist. Dieses Ziel soll durch den Einsatz von Assistenzsystemen und durch eine Erhöhung des Automatisierungsgrades erreicht werden.
Verkürzung der Interventionsprozesse, genaue Navigation durch optimierte Bildgebung (M²IBID), unmittelbare Lieferung der Gewebeproben an die Analytik und Applikation von Diagnostika und Therapeutika (M²oBiTE)
Arbeits- und Forschungsschwerpunkte von M²INT:
Erreichte Ziele:






Das Leitprojekt M²oBiTE, Molekulare Bioanalytik und Theranostika-Entwicklung, gliedert sich in die zwei Themenbereiche (A) Entwicklung einer Schnellanalytik für Patientenproben mittels massenspektrometrischer und spektroskopischer Bildgebung für eine individualisierte Therapieempfehlung basierend auf molekularer, ortsaufgelöster Gewebediagnostik und (B) Entwicklung und GMP-konforme (GMP = Good Manufacturing Practice) Herstellung innovativer Radiotheranostika für eine individualisierte, nicht-invasive Endoradiotherapie.
Ziel des ersten Themenbereichs ist die Verbesserung der Diagnostik und Klassifikation von Tumorgewebe im Hinblick auf definierte Therapieoptionen. Dazu werden die molekularen Fingerabdrücke der in Leitprojekt 1 gewonnenen Biopsate mit Hilfe massenspektrometrischer und spektroskopischer Methoden ortsaufgelöst im 3D-Raum analysiert. Für die Archivierung und Auswertung dieser molekularen Fingerabdrücke werden eine Datenbank für den systematischen Vergleich mit histopathologischen Daten entwickelt und neue Auswertungsmethoden implementiert.
Im zweiten Themenbereich werden zielgerichtete bekannte und neue Liganden sowie Antikörper und deren Derivate erforscht. Der Fokus liegt dabei auf Substanzen, die Therapieoptionen adressieren und die durch die molekulare Gewebeanalyse empfohlen werden können. In vitro und in vivo wird die Eignung der Tracer für die Bildgebung mittels NIR (Nahinfrarotspektroskopie) oder PET (Positronen-Emissions-Tomographie) und Endoradiotherapie evaluiert und anschließend die pharmakokinetischen Eigenschaften gegebenenfalls optimiert. Mittels aus Biopsaten gewonnenen 3D-Zellkulturen (Sphäroiden) wird in vitro die Toxizität der Radiotheranostika physiologisch realitätsnah analysiert. Hierzu wird zunächst eine automatisierte Gewebehomogenisation mit maximaler Zellausbeute etabliert.
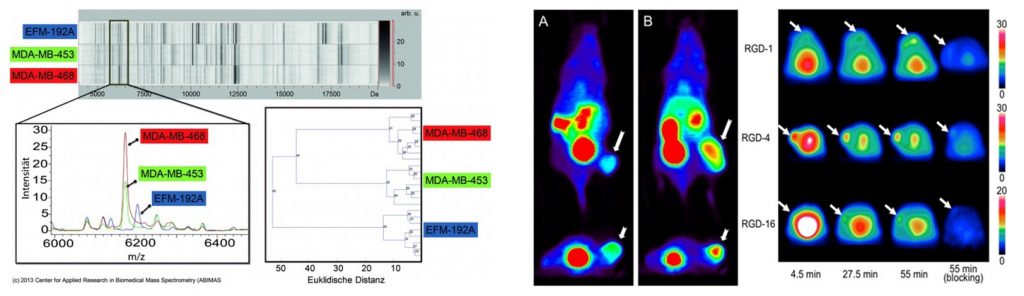
Um eine schnelle, hochflexible und GMP-konforme Herstellung der Radiotracer nach Bedarf zu ermöglichen, werden die manuellen Radiosynthesen für diagnostische und therapeutische Nuklide auf einem Synthesemodul oder zur manuellen Herstellung mittels eines sogenannten Markierungskits etabliert. Auf Basis der Biopsien und Biodistributionen wird eine patientenindividualisierte Behandlungsplanung für die Endoradiotherapie anhand von physiologisch basierten pharmakokinetischen Modellen implementiert, die die Auswahl der geeignetsten Therapeutika ermöglicht. In einer NIR-Tomographie-Plattform werden verschiedene Parameter optimiert, um die 3D-Biodistribution der NIR-Tracer präklinisch unter idealen Bedingungen zu evaluieren. Diese sollen in der klinischen Anwendung die (minimal)invasive Entfernung von Tumoren und Metastasen erleichtern und zusätzlich eine multimodale Bildgebung ermöglichen.
Integration der Gewebeanalyse und der Synthese von Radiotherapeutika in den Closed-Loop-Prozess von M²OLIE.
Arbeits- und Forschungsschwerpunkte von M²oBiTE:
Erreichte Ziele:





Das übergeordnete Ziel von Leitprojekt 3, Molekulare innovative Bildgebung für individualisierte Diagnostik, ist die Weiterentwicklung, Anwendung und Fusion mehrerer bildgebender Verfahren wie CT (Computertomographie), MRT (Magnetresonanztomographie) oder PET-CT (Positronen-Emissions-Tomographie-CT) für eine bilddatenbasierte und robotergestützte Intervention. Dadurch soll eine individualisierte Intervention eines krebserkrankten Patienten optimal und mit höchstmöglicher Präzision an einem Artis zeego® System durchgeführt werden können. Hierbei steht im Vordergrund, molekulare und neuartige funktionelle Bildinformationen, die mittels unterschiedlicher bildgebender Verfahren akquiriert werden, zu fusionieren und alle relevanten Informationen der unterschiedlichen Verfahren auf das Artis zeego® System zu transferieren. In diesem Prozess sollen dem an diesem System arbeitenden Arzt kompakt alle morphologischen, molekularen und funktionellen Informationen über den zu behandelnden Tumor patientenindividuell bereitgestellt und in Echtzeit mit den am Artis zeego® System (Cone Beam CT) akquirierten Daten zu multimodalen 3D- und 4D-Daten fusioniert werden.
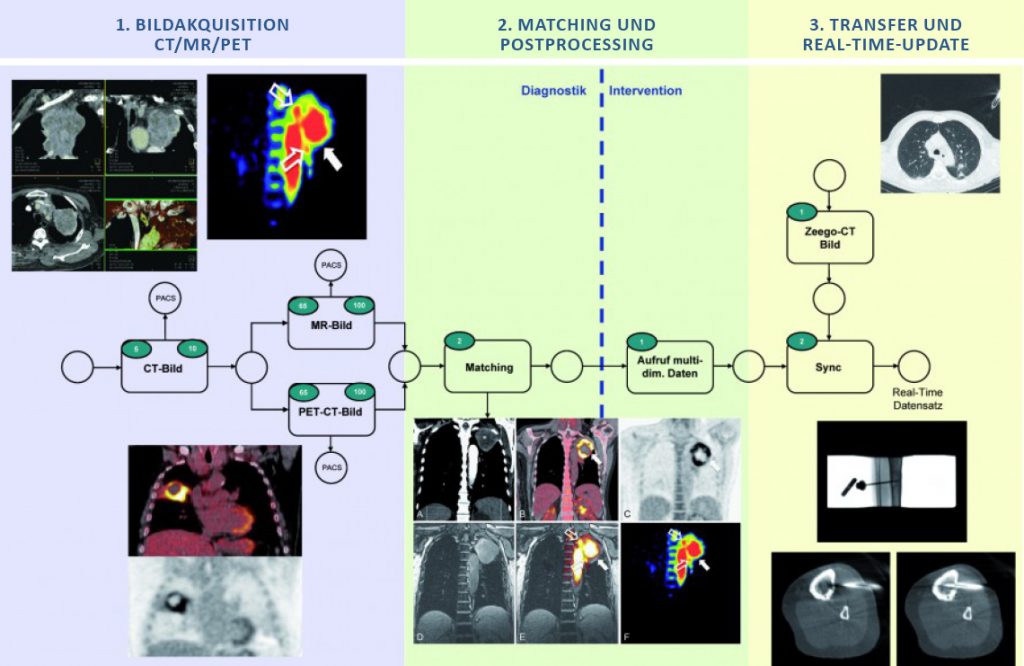
Optimale individualisierte Intervention mit höchstmöglicher Präzision bei einem krebskranken Patienten mit Hilfe eines Interventionsroboters an einem Artis zeego® System.
Arbeits- und Forschungsschwerpunkte von M²IBID:
Erreichte Ziele:








Das Querschnittsprojekt ProM²OLIE befasst sich mit einer organisationswissenschaftlichen Perspektive des molekularen Interventionsprozesses. Das Vorhaben hat das übergeordnete Ziel, durch ein holistisches Prozessmanagement und einen integrierten Ansatz den reibungslosen und wirtschaftlichen Gesamtablauf der verschiedenen Teilprozesse einer molekularen Behandlung von Patienten sowohl während der Forschungsphase als auch im späteren Realbetrieb sicherzustellen. ProM²OLIE operationalisiert das Ziel von M²OLIE, den Patienten in einem sogenannten One-Stop-Shop mit den Prozessschritten Diagnose, Analyse, Vorbereitung und Intervention innerhalb von wenigen Stunden zu behandeln. Dazu werden folgende Maßnahmen durchgeführt:
Erreichte Ziele



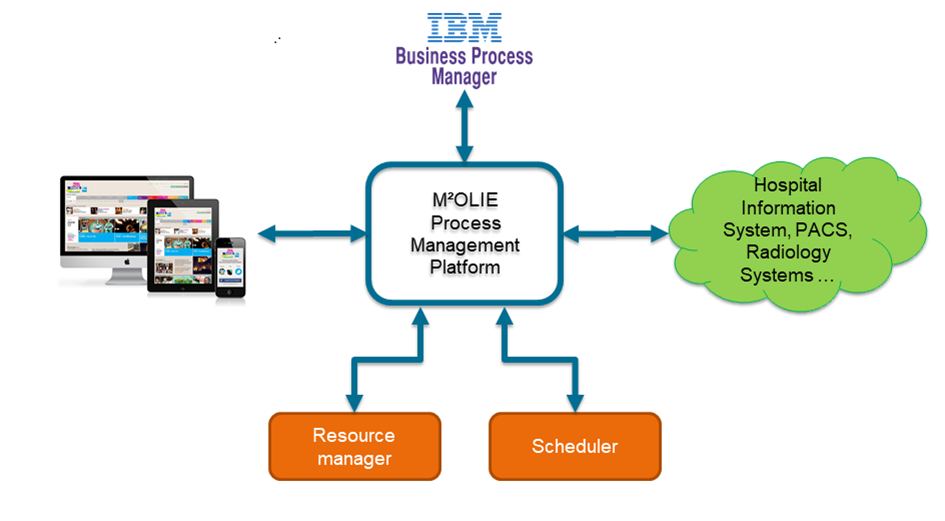
Das Querschnittsprojekt M²PROCIT, Mannheim Molecular Process IT-Platform, befasst sich mit der Entwicklung eines integrierten, adaptiven Workflow-Management-Systems (WFMS) für die ganzheitliche Digitalisierung des molekularen Interventionsprozesses.
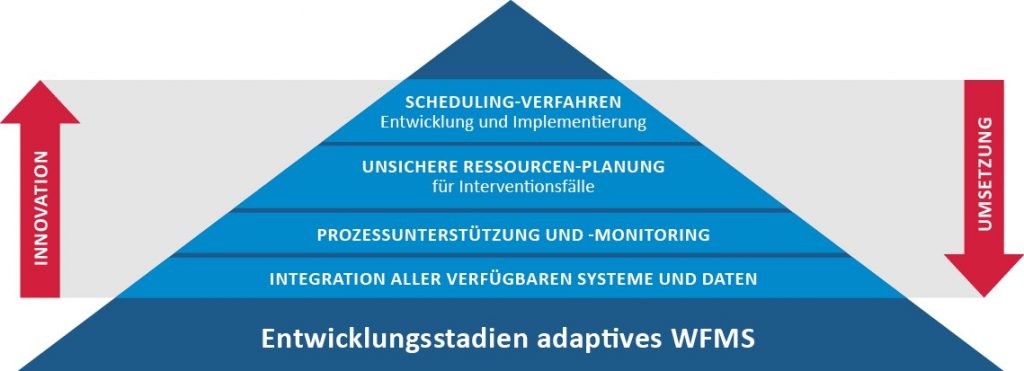
Herzstück und größte Herausforderung von M²PROCIT ist es, den One-Stop-Shop (molekularen Interventionsprozess) in ausführbaren Prozessmodellen abzubilden, deren Ausführung in einem Prozessmanagementsystem durch die Integration der situativ relevanten Datenquellen zu ermöglichen und schließlich durch die Bereitstellung von Komponenten zur Ressourcenplanung und der Ausführungsplanung für Teilprozesse („Scheduling“) einen kostenoptimierten Betrieb zu gewährleisten. Der One-Stop-Shop beinhaltet die taggleiche Diagnose, Analyse, Vorbereitung und Intervention für alle eintreffenden Patienten für M²OLIE. Hierdurch entsteht ein komplexer und dynamischer Prozess, welcher bislang in traditionellen klinischen Prozessmanagementsystemen nicht abgebildet werden kann. Der One-Stop-Shop bedingt, dass die aufeinanderfolgenden Prozessschritte ad hoc und flexibel determiniert werden müssen. Dadurch geht ein hoher Grad an Vorplanungssicherheit verloren, und Erkenntnisse über die direkt im nächsten Prozessschritt benötigten Ressourcen kristallisieren sich erst kurzfristig und sequentiell heraus.
Die unter den oben skizzierten Vorgaben entwickelte M²OLIE Process Management Plattform übernimmt das Management der einzelnen Komponenten und führt die benötigten Informationen zusammen.