
Die eindeutige Identifikation und Charakterisierung therapierelevanter Herdbefunde mit innovativen diagnostischen Methoden stellt die Grundlage zur minimalinvasiven Therapie (u. a. Radionuklidtherapie) für den Patienten mit Oligometastasen im Closed-Loop-Prozess von M²OLIE dar. Sie erlaubt außerdem eine Prädiktion des erwarteten Therapieansprechens und die frühzeitige Erkennung von Patienten, die nicht auf eine bestimmte Therapie ansprechen (Non-Responder). Ziel des Verbundvorhabens M²IBID ist es, diese Grundvoraussetzungen für die Festlegung einer effektiven Therapie im Sinne der „Präzisionsmedizin” zu erforschen. Die Techniken der diagnostischen und interventionellen Bildgebung und molekularen massenspektrometrischen (MS) Bioanalytik sollen tiefer integriert und klinisch evaluiert werden. Das innovative Forschungsziel liegt in der idealen Verknüpfung all dieser Informationen, um dann neuartige Tumorcharakterisierungen mittels Radiomics-Ansätzen und Techniken des maschinellen Lernens zu erforschen, welche die patientenindividuelle Intervention verbessern.
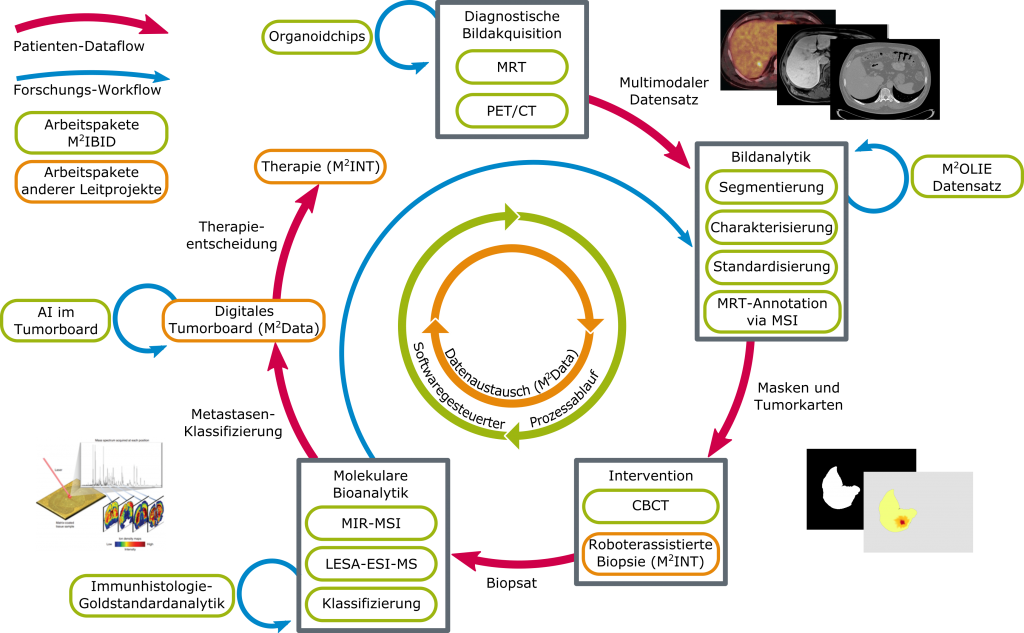
Ziel in Teilvorhaben 1 (Leitung: Universität Heidelberg) „Molecular and Multimodal Imaging and Processing (M²IP)“ ist die Entwicklung und Optimierung einer standardisierten multimodalen diagnostischen und interventionellen Bildgebung für den oligometastasierten Patienten. Im Bereich der diagnostischen Bildgebung werden hochaktuelle, innovative Akquisitionstechniken (z. B. MR-Fingerprinting, MR-Elastographie, MR-Perfusion, MR-Diffusion, multimodales Atemtracking) und diagnostische PET-Tracer entwickelt und evaluiert.








Das Teilvorhaben 2 (Leitung: Hochschule Mannheim) „M²OLIE Tumor- und Theranostika-Analytik“ (M²OT²AN) adressiert mehrere zentral wichtige Teilaspekte des Gesamtvorhabens „Molekulare innovative Bildgebung für individualisierte Diagnostik“ (M²IBID). Der Fokus liegt dabei i) auf der Erforschung automatisierter Methoden der molekularen Bioanalytik von Tumoren und Metastasen, ii) der Erforschung neuartiger Bildverarbeitung zur Extraktion multimodaler Imaging-Biomarker, iii) deren Verknüpfung mit radiologischer Bildanalytik im M²OLIE Closed-Loop-Prozess zu einer integrierten radiologischen und molekularen Diagnostik für die individualisierte Therapieentscheidung und schließlich iv) der Erforschung der Nutzung von Organoiden für die Untersuchung von Theranostikaeigenschaften.




Ziel des Teilvorhaben 3 (Leitung: DKFZ) SIM²BA ist es, den integrierten Workflow und seine klinische Evaluation zu unterstützen, um potentielle Interventionstargets (primäre und sekundäre Lebertumore) für die lokale, perkutane minimalinvasive als auch für die dezidierte, systemische nichtinvasive Radionuklidtherapie zu identifizieren und charakterisieren. Dieser Workflow zeichnet sich durch die Verknüpfung aller Informationen, die mithilfe der diagnostischen Bildgebung als auch der molekularen Bioanalytik gewonnen werden können, aus. Die eindeutige Identifikation und Charakterisierung therapierelevanter Herdbefunde mit innovativen diagnostischen Methoden und besonders ihrer Integration und Kombination (Radiomics) stellen die Grundlage zur minimalinvasiven Therapie für den oligometastasierten Patienten dar.



Das Teilvorhaben 4 (Leitung: Mint Medical GmbH), M²KoRaMo, stellt den Anknüpfungspunkt zwischen den für M²OLIE charakteristischen und namensgebenden molekulardiagnostischen Verfahren sowie bildbasierenden Verfahren dar. Dadurch wird die Integration der molekulardiagnostischen Ansätze in das Patientenmodell ermöglicht. Unterschiedliche bildgebende Verfahren müssen mit molekularbiologischen Verfahren in Verbindung gebracht werden, um eine individualisierte Intervention eines oligometastasierten Patienten mit höchstmöglicher Präzision sowie maximal schonend durchzuführen. Damit die hierdurch erarbeiteten diagnostischen Parameter keine Insellösungen verbleiben, ist eine Rückführung in den klinischen Prozess und in eine radiologische Datenanalyse-Plattform unbedingt erforderlich.



